El treball publicat a Nature Methods avalua les eines utilitzades per detectar el genoma del càncer
Eduard Porta-Pardo, un investigador sènior del departament de ciències de la vida al Barcelona Supercomputing Center – Centro Nacional de Supercomputación (BSC-CNS), amb la col·laboració d’un equip internacional de científics, ha realitzat la primera anàlisi comparativa dels algoritmes subgenètics que exploten la informació genètica de les bases de dades de càncer. Aquestes potents eines de filtratge de dades són útils a l’hora de descabdellar la complexitat del càncer i trobar mutacions no identificades prèviament però que són importants en la formació de cèl·lules canceroses.
L’estudi, publicat avui a Nature Methods, revisa, classifica i descriu els punts forts i febles de més de 20 algoritmes desenvolupats per grups de recerca independents. L'avaluació dels mètodes d'anàlisi del genoma del càncer és clau per a la selecció de les estratègies més adequades que s’integraran en la plataforma de medicina personalitzada del BSC.
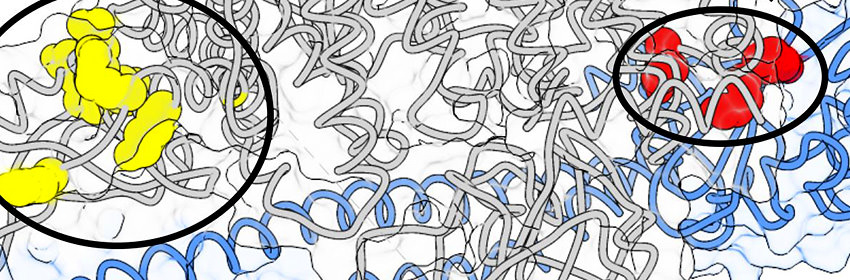
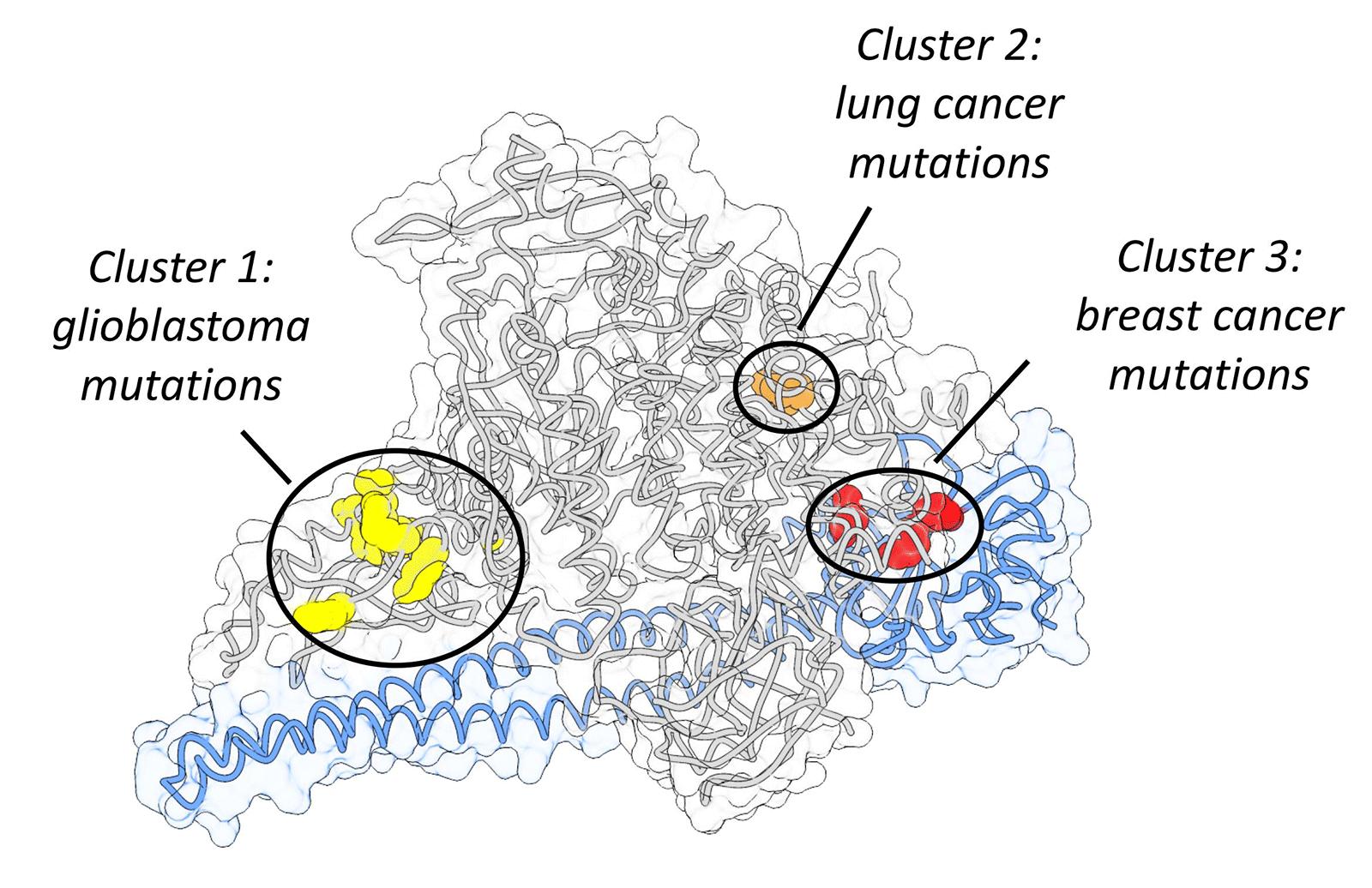
Tot i la disponibilitat creixent de seqüències genòmiques, una creença comuna és considerar un gen com una sola unitat. No obstant, hi ha una sèrie d'esdeveniments, com ara substitucions, duplicacions i pèrdues d’ADN que es poden produir dins d'un gen, és a dir a nivell sungenètic. Els algoritmes subgenètics proporcionen una visió d'alta resolució que pot explicar per què diferents mutacions en el mateix gen poden provocar fenotips diferents depenent de com la mutació afecta regions proteiques específiques. Un bon exemple de com les mutacions subgenètiques influencien el càncer és el gen NOTCH1. Les mutacions en certes regions de NOTCH1 fan que actuï com un supressor tumoral en els càncers de pulmó, pell, cap i coll. En canvi, les mutacions en una altra regió poden promoure la leucèmia limfocítica crònica i la leucèmia limfoblàstica aguda de cèl·lules T. Per tant, és incorrecte suposar que les mutacions en un gen tindran les mateixes conseqüències independentment de la seva ubicació.
Els investigadors de l'estudi van aplicar cada algoritme subgenètic a les dades procedents de l’Atlas del Genoma del Càncer (TCGA), un conjunt de dades a gran escala que inclou informació de genomes de 33 tipus de tumors de més de 11.000 pacients. "El nostre objectiu no era determinar quin algoritme funcionava millor que un altre, ja que això dependrà de la pregunta que es formula", diu Eduard Porta-Pardo, primer autor de l’article. "En canvi, sí que volem informar als possibles usuaris sobre com les diferents hipòtesis darrere de cada algoritme subgenètic influeixen en els resultats i com els resultats difereixen dels mètodes que funcionen a nivell de gen sencer". Porta Pardo és un ex-investigador postdoctoral del Sanford Burnham Prebys Medical Discovery Institute (SBP) que recentment s’ha incorporat al departament de ciències de la vida del BSC sota la direcció d'Alfonso Valencia, també coautor d'aquest treball.
Els investigadors han arribat a dos descobriments importants. En primer lloc, han descobert que els algoritmes són capaços de reproduir la llista de gens del càncer coneguts, validant l'enfocament subgenètic i el vincle entre aquests gens i el càncer. En segon lloc, han trobat una sèrie de nous gens directors del càncer, que estan implicats en el procés d'oncogènesi i que no van ser detectats amb els enfocaments del gen sencer.
“Trobar nous gens directors del càncer és un objectiu rellevant en l'anàlisi del genoma del càncer", afegeix Porta-Pardo. Aquest estudi hauria d'ajudar els investigadors a comprendre els avantatges i inconvenients dels algoritmes subgenètics utilitzats per trobar potencials noves dianes de fàrmacs per al tractament del càncer.
Tot i que el Sanford Burnham Prebys Medical Discovery (SBP) ha liderat el projecte, l’article ha estat l'esforç entre institucions internacionals com la Harvard Medical School, el Barcelona Supercomputing Center, l'Institut de Recerca Biomèdica de Barcelona (IRB), la Universitat Pompeu Fabra (UPF) i el Centro Nacional de Investigaciones Oncológicas (CNIO), entre altres.
Treball de Referència
Porta-Pardo E, Kamburov A, Tamborero D , Pons T, Grases D, Valencia A, Lopez-Bigas N, Getz G and Godzik A (2017). Comparison of algorithms for the detection of cancer-drivers at sub-gene Resolution. Nature Methods. dx.doi.org/10.1038/nmeth.4364